Please select your location and preferred language where available.
当社はSSDを活用した高速な顕微鏡画像処理技術を開発しています*。その成果の1つである、神経細胞の電位を顕微鏡動画から実時間で抽出する手法[1]を紹介します。
脳では神経細胞(ニューロン)が互いに電気信号をやり取りしており、個々の神経細胞の電位を観測し脳活動を調べることは、脳の仕組みを理解する重要な手段のひとつです。従来は、ネズミ等の実験動物の神経細胞に針やピペットを接触させて電位を計測していましたが、多数の細胞を同時に観測することが困難な上、脳に異物を挿入しダメージを与えてしまう問題がありました。これに対し、近年ではボルテージ・インジケータと呼ばれる蛍光物質を設計して、神経細胞の電位を光に変換して顕微鏡で撮影するボルテージ・イメージングと呼ばれる手法の開発が進んでいます[2]。
端的に言うと、活動している神経細胞は点滅しているように写ります。これにより、頭蓋骨に穴を開けるなど脳環境への影響は依然としてあるものの、観測対象となる細胞の周辺組織を傷つけない侵襲性の低い方法により複数の神経細胞の観測が可能となります。しかし、神経細胞のミリ秒単位の電位変化を捉えるために毎秒数百から千フレームの高速撮影を行うため、10秒程度の撮影でも動画データサイズは数GBとなり、データ処理が課題となっていました。
当社が協力していた複数大学の脳科学研究者によると、動物実験の条件を変えながら1日当たり合計3-4時間の撮影を行うのが望ましいものの、結果として得られた1TB程度の動画データを処理するのに数日かかってしまうために小規模の実験に留めざるを得ないとのことでした。実験の規模を大きくしても処理が追いつかず、データが単に蓄積していくだけとなってしまうからです。
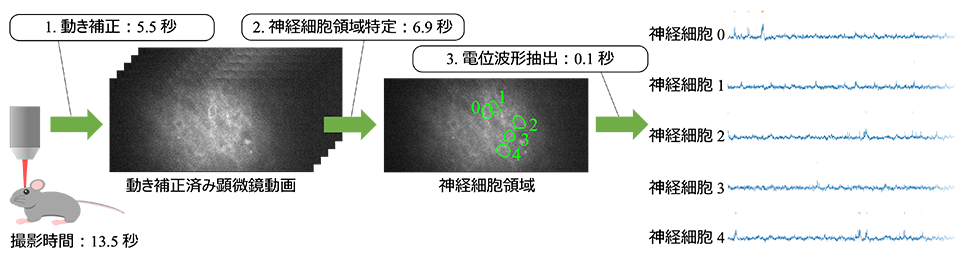
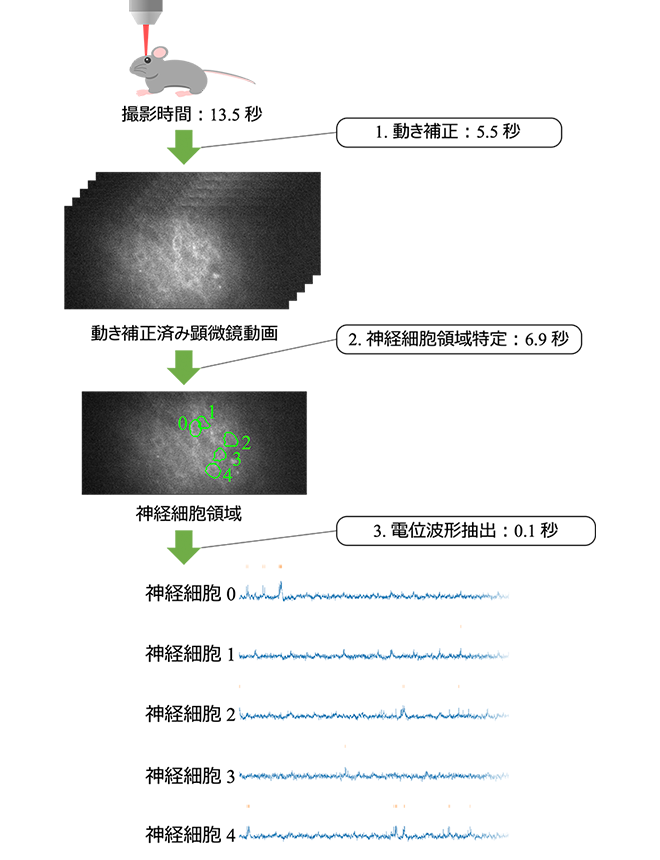
そこで当社は、顕微鏡動画から神経細胞電位を抽出するデータ処理を高速化し、動画を撮影するのにかかった時間以下の時間での処理を実現しました。顕微鏡撮影と等速度で処理できる実時間性能となるため、処理されず蓄積されるだけのデータがなくなります。データ処理は具体的には、以下の3ステップからなります(図1)。
- 動き補正:動物は頭部を器具で固定しますが、それでも微小な動きが顕微鏡下では無視できない大きさとなって観測されるためそれを画像処理で相殺します。結果として、静止した神経細胞が点滅する動画が得られます。
- 神経細胞領域特定:補正した動画から神経細胞を検出し、各神経細胞が占める画像上の領域を特定します。
- 電位波形抽出:特定した個々の神経細胞の領域から電位波形を抽出します。
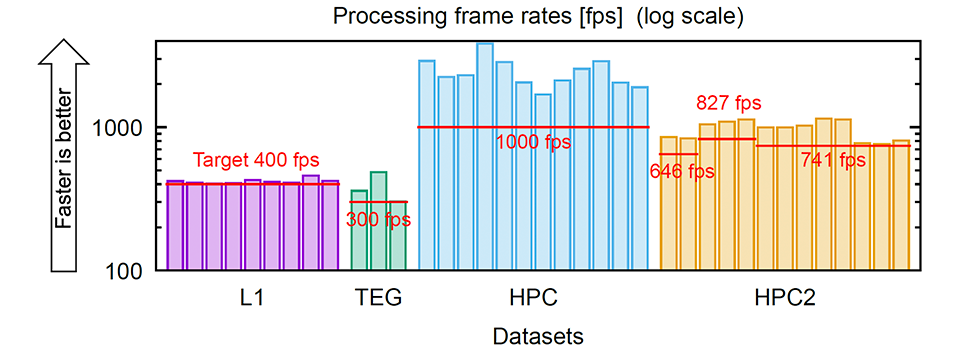
図1中の例は、毎秒741フレームの撮影速度で1万フレーム撮影した13.5秒の動画です。これを5.5秒で動き補正し、6.9秒で神経細胞の領域(この例では5つ)を特定し、0.1秒で各細胞の電位波形を抽出することができました。合計で12.5秒の処理時間となり、動画の撮影時間を下回る(撮影速度を上回る)実時間性能となっています。
ステップ1は、よく知られた既存手法を活用した入念なGPU (graphics processing unit)実装により高速化しています。具体的には、画像を複数のパッチに分割してGPUで並列処理する、フレーム間の類似度をZNCC (zero-mean normalized cross-correlation)指標で評価する際に必要な画素値の総和をテーブル参照により高速化[3]するなどです。
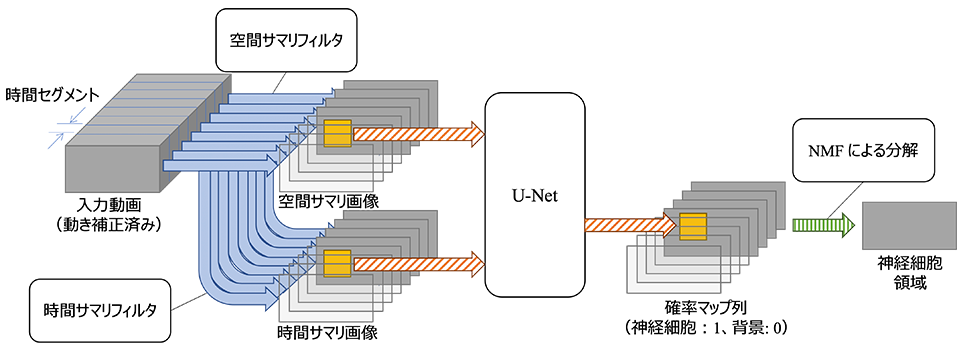
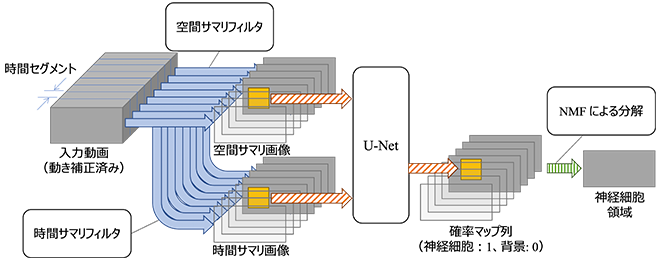
ステップ2のアルゴリズムを図2に示します。まず、動き補正済みの入力動画を時間方向に一定長(50フレーム)に区切った時間セグメントを、サマリフィルタで要約します。50フレームの時間セグメントが、空間的特徴および時間的特徴を捉える2枚のサマリ画像に要約されるためフレーム数が大幅に減ります。さらに、サマリ画像の組から複数のパッチを取り出して、領域抽出用の畳み込みニューラルネットワークであるU-Net[4]を用いて、各画素が神経細胞か否かを示す確率を出力します。U-Netの学習はボルテージ・イメージングの顕微鏡撮影過程をシミュレーションした人工画像により行いました。
一旦学習が済めば、U-Netの動作は高速です。U-Netが出力するパッチを統合することで、各時間セグメントに対応する神経細胞の確率マップが得られます。最後に、確率マップ列を非負行列分解(NMF, nonnegative matrix factorization)を用いて、神経細胞領域とそれらの時間変化の積に分解します。
ステップ3は、各神経細胞の領域に含まれる画素値を毎フレーム平均するのみの簡単な処理であり、単純な並列化により十分に高速な処理が可能です。
以上により、表1に示す複数のデータセット群からなる37本の顕微鏡動画全てに対して、各動画の撮影フレームレートを上回る速度での処理を実現しました(図3)。これはボルテージ・イメージング向けデータ処理の既存手法であるSGPMD-NMF[5]の100倍以上、VolPy[6]の4-8倍高速な処理速度となっており、単一のデスクトップPC(仕様は表2参照)で実時間処理を達成する初の手法となっています。コードはGitHubに公開されています。
※表を左右にスクロールすることができます
|
データセット群 |
動物 |
脳領域 |
フレームレート (fps) |
動画数 |
ボルテージ・インジケータ |
|---|---|---|---|---|---|
|
L1 |
ネズミ |
皮質第一層 |
400 |
9 |
Voltron[7] |
|
TEG |
ゼブラフィッシュ |
被蓋 |
300 |
3 |
Voltron[7] |
|
HPC |
ネズミ |
海馬 |
1,000 |
12 |
paQuasAr3-s[8] |
|
HPC2 |
ネズミ |
海馬 |
645 – 826 |
13 |
SomArchon[2] |
|
Component |
Specification |
|---|---|
|
CPU |
AMD Ryzen Threadripper 3960X (24 cores, 3.8 GHz) |
|
RAM |
DDR4 3200 MHz, 192 GB (6 channels x 32 GB) |
|
GPU |
2x NVIDIA GeForce RTX 2080 Ti |
|
SSD |
KIOXIA EXCERIA PRO 2 TB (NVMe™, PCIe® Gen 4 x4) |
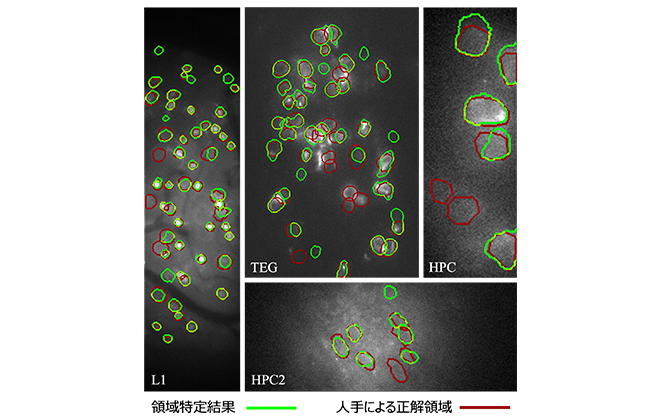
提案手法による神経細胞領域特定の精度を、データセットに付属の正解データと比較したところ平均で80%程度の一致を示しました。図4に例を示します。これは従来手法に比べて同程度または良い精度です。正解データは、複数人が人手で入力した領域を、全員が合意に至るまで相互チェックしながら修正して作成されたものです。高速撮影したノイズの多い画像のため、個人の初期判断は合意後の正解データに対して80%程度しか一致しません。そのため、80%の神経細胞領域特定精度とは、一人の人間の手作業を置き換える程度の精度であると見なすことができます。
研究者の想定と異なる出力となった細胞領域の修正や、抽出した電位波形のさらなる分析など、提案手法がカバーしないデータ処理は残りますが、GPUとSSDを搭載したコンピュータが1台あれば、撮影された多数の神経細胞の大部分について電位波形を即座に提示できることは、脳科学研究者にとって有用であり、ボルテージ・イメージングを用いた脳科学研究の加速につながると期待しています。当社はこれからも、SSDおよびメモリ技術を活用した高速データ処理により先端研究に貢献していきます。
本成果は国際学会BIBM 2023で発表されました。
NVMeは、NVM Express, Inc.の米国またはその他の国における登録商標または商標です。
PCIe は、PCI-SIG の登録商標です。
その他記載されている社名・商品名・サービス名などは、それぞれ各社が商標として使用している場合があります。
文献
[1] Y. Bando et al. Real-time neuron segmentation for voltage imaging. IEEE International Conference on Bioinformatics and Biomedicine (BIBM), 813–818, 2023.
[2] K. D. Piatkevich et al. Population imaging of neural activity in awake behaving mice. Nature 574, 413–417, 2019.
[3] J. P. Lewis. Fast template matching. Vision Interface, 120–123, 1995.
[4] O. Ronneberger, P. Fischer, and T. Brox. U-Net: Convolutional networks for biomedical image segmentation. Medical Image Computing and Computer-Assisted Intervention (MICCAI), 234–241, 2015.
[5] M. E. Xie et al. High-fidelity estimates of spikes and subthreshold waveforms from 1-photon voltage imaging in vivo. Cell Reports 35(1), 108954, 2021.
[6] C. Cai et al. VolPy: Automated and scalable analysis pipelines for voltage imaging datasets. PLoS Computational Biology 17(4), e1008806, 2021.
[7] A. S. Abdelfattah et al. Bright and photostable chemigenetic indicators for extended in vivo voltage imaging. Science 365(6454), 699–704, 2019.
[8] Y. Adam et al. Voltage imaging and optogenetics reveal behaviour-dependent changes in hippocampal dynamics. Nature 569, 413–417, 2019.

